|
Mapping three-dimensional intratumor proteomic heterogeneity in uterine serous carcinoma by multiregion microsampling
Allison L. Hunt, Nicholas W. Bateman, Waleed Barakat, Sasha C. Makohon-Moore, Tamara Abulez, Jordan A. Driscoll, Joshua P. Schaaf, Brian L. Hood, Kelly A. Conrads, Ming Zhou, Valerie Calvert, Mariaelena Pierobon, Jeremy Loffredo, Katlin N. Wilson, Tracy J. Litzi, Pang-Ning Teng, Julie Oliver, Dave Mitchell, Glenn Gist, Christine Rojas, Brian Blanton, Kathleen M. Darcy, Uma N. M. Rao, Emanuel F. Petricoin, Neil T. Phippen, G. Larry Maxwell & Thomas P. Conrads
Clinical Proteomics volume 21, Article number: 4 (2024)
作者调查了子宫浆液性癌 (USC) 肿瘤微环境中的三维异质性。 他们使用空间分辨激光显微切割 (LMD) 从九名患者的肿瘤组织样本中选择细胞亚群。
块通过冷冻切片机切成连续的 10 µm 组织切片,并使用 LMD 在标本块的 5 个独立的空间不同层次中分离富集的肿瘤上皮 (ET) 或肿瘤相关基质 (ES) 细胞。 LMD 组织经过胰蛋白酶消化,每个样品的 5 µg 胰蛋白酶肽段用异丙醇 16 重串联质量标签标记。在 LC-MS/MS 之前,样品通过碱性反相液相色谱分离。蛋白质水平丰度根据对应于单个蛋白质登录号的至少两个 PSM 的 TMT 报告离子比率计算。
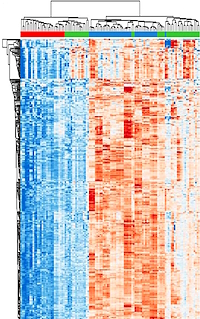
对每个患者特有的平均 9548±450 个蛋白质进行了定量分析,其中在所有九名患者中共同定量分析的蛋白质总共有 6503 个。 对 351 种差异蛋白质(绝对中位差 >1)进行无监督层次聚类分析,揭示了两个主要分支,其中 ET 样本与 ES 样本独立聚类。反相蛋白微阵列数据显示有 160 种蛋白质与 MS 共同定量。
他们还将本 USC 研究中 ET 和 ES 之间显著改变的蛋白质与他们之前卵巢癌数据集中的蛋白质进行了比较。 他们发现总共有 313 种蛋白质发生共同改变,并在 ET 和 ES 之间表现出相同的丰度趋势。
|
|