 |
|
欢迎
欢迎加入 我们在圣安东尼奥ASMS大会期间举办的早餐会议,时间:6月6日至7日。
如果您需要研究含有硒代半胱氨酸(selenocysteine)的蛋白,我们可以给您提供一些建议。
这个月出版的特色文章中,作者利用核糖体分析以及蛋白组学技术鉴定了细菌中表达基因的翻译起始位点。如果您最近也有文章发表并且希望我们列举在下一期的Newsletter中,
请发给我们
相关的PDF 或者URL.
本月Mascot小技巧告诉您database *. stats file 的重要性。
如果你有任何建议和问题,请随时 联系我们。 |
|
|
|
 |
 |
 |
|
加入我们——圣安东尼奥ASMS大会
在6月即将到来的圣安东尼奥ASMS大会上,我们诚挚邀请您参加我们的早餐会议
。我们将举办两场报告,您可以从中了解数据库搜索和定量的最新应用和进展。早餐会议免费,但是需要提前注册。
请点击此处进行注册并获取会议详情
6月6日(周一): 7:00 am - 8:00 am
Ancient proteomes - proteomics in archaeology, palaeontology and forensics
报告人: Michael Buckley(曼彻斯特大学,Manchester University)
Mass tolerant and error tolerant searches: how do they compare?
报告人: Matrix Science
6月7日(周二): 7:00 am - 8:00 am
From Samples to Biomarker Discovery: The keys for Large Scale Proteomics
报告人: John Corthesy(雀巢健康科学研究院 Nestlé Institute of Health Sciences)
New features in Mascot Distiller - MSE, MS3 reporter ion quant, and more
报告人: Matrix Science
|
 |
 |
 |
 |
|
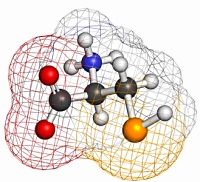
Selenocysteine(硒代半胱氨酸)和U
硒代半胱氨酸Selenocysteine (U)是一种罕见的残基,但是在人类蛋白组数据库中中,该残基出现了46次。如果您恰巧正在研究硒蛋白P(selenoprotein P)——该蛋白含有10个硒代半胱氨酸残基——你必须对该残基同时设定修饰和非修饰形式。因为该残基信息刚刚加入Unimod中,你可能无法在Mascot Server configuration editor中查询到。该 博文 告知您如何更新您本地的Unimod 文件。
Unimod更新后,你会发现搜索表格中列出了硒代半胱氨酸上的修饰形式,同时还包含了其他您已经在本地定义的修饰:
- Carbamidomethyl (U)
- Carboxymethyl (U)
- Dioxidation (U)
- MolybdopterinGD (U)
- Oxidation (U)
|
 |
 |
 |
 |
|
关于 Matrix Science
Matrix Science 为蛋白组学的研究人员以及科学家提供生物信息分析工具,帮助他们更快速,更可信的鉴定和定量蛋白。Mascot 软件全线支持来自Sciex,
Agilent, Bruker, Shimadzu, Thermo Scientific 以及 Waters质谱仪生成的质谱数据。
请联系康昱盛以获取更多的信息。
|
 |
 |
|