|
An affinity-directed phosphatase, AdPhosphatase, system for targeted protein dephosphorylation
Luke M. Simpson, Luke J. Fulcher, Gajanan Sathe, Abigail Brewer, Jin-Feng Zhao, Daniel R. Squair, Jennifer Crooks, Melanie Wightman, Nicola T. Wood, Robert Gourlay, Joby Varghese, Renata F. Soares, and Gopal P. Sapkota
Cell Chemical Biology 30, 188–202, February 16, 2023
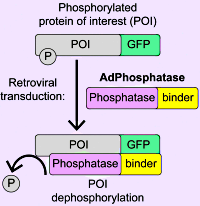
作者开发了一种名为AdPhosphatase的新方法,该方法可以选择性、高效地靶向结合GFP标记的蛋白质进行去磷酸化。AdPhosphatase分子由纳米抗体和磷酸酶催化亚基(PPP1CA/PPP2CA)结合而成。
他们比较了AdPhosphatase与小分子激酶抑制剂的去磷酸化效果,发现两种方法对脱磷酸化的作用程度相当。
使用TMT标记定量和磷酸肽富集分析,鉴定出11,821条特异的磷酸化肽段。其中,21种蛋白质对应的肽段磷酸化水平被AdPhosphatase显著下调了2倍以上。此外,AdPhosphatase介导的脱磷酸化特异性非常高,只有一小部分非靶向磷酸化肽段被显著下调。
|
 |